1.Sequencing 분석 기술
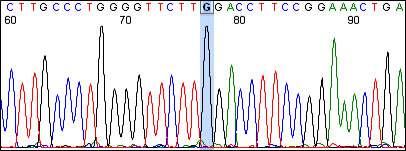
PCR 산물 및 vector 등의 DNA를 정량하여 BigDye Terminator로 반응 시킨 후 ABI PRISM 3130 DNA Analyzer 기기를 사용하여 분석하는 방법이다. 분석 항목에 제시된 모든 유전자에 대해 sequencing이 가능하고, 분석 항목에 제시된 유전자가 아니라 해도 기술 개발을 통한 Sequencing이 가능하다.
 |
 |
| < 3130 Genetic Analyzer > |
< Sequencing 결과 > |
2. Multiplex SNaPshot 분석 기술
ABI PRISM 3130 DNA Analyzer와 ABI SNaPshot multiplex kit를 사용하여 Single base pair primer extension assay의 원리를 이용한 고속 유전형 분석 방법이다. SNaPshot 분석법은 multiplex 분석이 가능하기 때문에 타 유전형 분석법에 비하여 정확도, 시간, 비용면에서 탁월한 장점이 있는데, genomic DNA 분리 및 PCR 과정 후 SNaPshot 반응 및 분석은 대략 4시간 정도의 시간이 소요된다. 한번의 실험으로 96개의 샘플을 실험할 수 있고, 한 샘플 당 11개의 SNP 분석이 가능하다. 이 분석법으로 유전형 분석이 가능한 유전자는 CYP1A2, CYP2C8, CYP2C9, CYP2C19, CYP3A4, CYP3A5, CYP2D6, CYP2B6, CYP2A6, VKORC1, PXR 등이 있다.
 |
 |
| < 3130 Genetic Analyzer > |
< SNaPshot 결과> |
3. TaqMan을 이용한 분석 기술
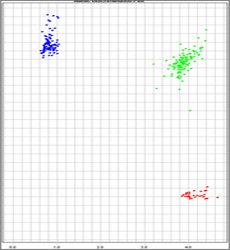
Taqman assay 방법으로 하루 1200개 이상의 샘플을 ABI 7900 HT Analyzer를 사용하여 고속 유전형 분석을 할 수 있다. 소량의 sample로도 분석이 가능하며 PCR contamination으로 인한 잘못된 결과를 방지할 수 있다. 또한, 기존의 PCR 방법에 사용된 primer 쌍에 probe가 추가된 3개의 탐침자(oligo)로 PCR을 수행하게 되어 결과의 정확성이 탁월하다. 한 번의 실험으로 384개의 샘플을 실험할 수 있고, 한 샘플 당 1개의 SNP이 분석이 가능하다. 한번에 다량의 샘플을 분석할 수 있어 비용이 절감되는 장점이 있다. genomic DNA 분리 후 Taqman 반응 및 분석은 대략 3시간 정도의 시간이 소요된다. ABI 및 roche 의 회사의 시판되는 모든 Taqman probe에 대해서 분석 가능하다.
 |
 |
| < Real time PCR > |
< Taqman 결과> |
4. HRM (High Resolution Melting) 방법을 이용한 분석 기술
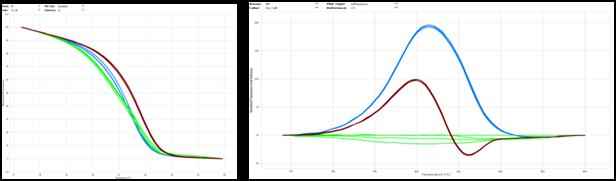
Illumina Eco사의 Real time PCR 장비를 이용한, HRM 기법은 실시간 연쇄중합 반응 (real time PCR) 기법 중 비교적 최신 기법으로 PCR 산물이 DNA 서열에 따라 고유의 Tm(melting temperature)를 갖는 것을 이용한 SNP 분석 기법이다. PCR 산물이 만들어질 때 DNA 이중 가닥에 끼어 들어가는 intercalating dye를 첨가하게 되면 PCR 산물에 끼어 들어가 형광 파장을 방출하게 되는데 이때 온도를 낮은 온도에서 높은 온도로 서서히 올리면 PCR 산물은 고유의 Tm값에서 변성(denaturation), 즉 이중가닥에서 단일 가닥으로 바뀌게 되고 형광 값이 급격히 떨어지게 된다. 이때의 온도를 melting temperature라 하며 이 melting temperature의 차이를 구분하여 PCR 산물에서 서열 하나의 차이까지 구분해내는 기법이다. HRM 기법은 기존의 taqman 분석 기법에 비해 프로브(probe) 없이 PCR 프라이머만 사용하므로 실험 단가적인 부분이 절감될 수 있을 뿐만 아니라 프로브에 비해 셋팅이 용이한 장점이 있다. 또한 기존의 시퀀싱(sequencing), SNaPshot 분석, 파이로시퀀싱(Pyrosequencing)에 비해 PCR 후 과정이 없이 바로 분석이 가능하므로, 시간적인 면에서도 효율적인 방법이라 할 수 있다. 한번의 실험으로 48개의 샘플을 90분 이내에 실험할 수 있다. 이 분석법으로 유전형 분석이 가능한 유전자는 CYP2C9, CYP2C19, CYP3A4, CYP3A5, CYP4F2, UGT1A1, MDR1, VKORC1 등이 있다.
 |
 |
| < Illumina Eco > |
< HRM 결과> |
5. Pyrosequencing를 이용한 분석 기술
Biotage사의 PSQ MA96 자동화 DNA sequencer 장비를 이용하고, enzyme-cascade를 사용하여 4개의 enzyme과 substrate를 사용하여 DNA template에 상보적으로 하나의 nucleotide가 incorporation 될 때마다 빛이 발생하도록 하여, 합성을 해 나가면서 DNA sequencer를 정확하고 빠르게 분석하는 방법이다. 한 번의 실험으로 96개의 샘플을 실험할 수 있고, 한 샘플당 3개의 SNP이 분석이 가능하다. genomic DNA 분리 및 PCR 과정 후 Pyrosequencing 반응 및 분석은 대략 20분 정도의 시간이 소요되는데, 타유전형 분석법에 비하여 시간적이 절감이 탁월한 분석법이다. 이 분석법으로 유전형 분석이 가능한 유전자는 CYP2B6, CYP2C8, CYP2C9, CYP2C19, CYP3A4, CYP3A5, CYP2D6, UGT1A1, UGT1A9, TPMT, HNF4A, MDR1, BCRP, MRP2, OCT1, OCT2, OATPC, ALDH2, ADH1B, MTHFR, VKORC1등이 있다.
 |
 |
| < Pyrosequencer > |
< Pyrosequencing 결과> |
6. PCR-RFLP (Restriction Fragment Length Polymorphism) 방법을 이용한 분석 기술
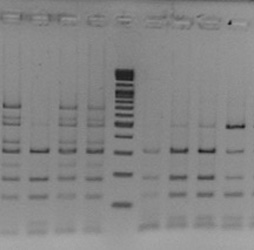
각 개인으로부터 얻은 DNA를 적당한 제한효소로 자르고 전기영동을 한 후 나타나는 절편 크기를 확인하여 유전형을 결정하는 방법이다. 한 번의 실험으로 1개의 샘플을 실험할 수 있고, 한 샘플 당 1개의 SNP이 분석이 가능하다. Genomic DNA 분리 및 PCR 후 RFLP 반응 및 분석은 대략 5시간 정도의 시간이 소요된다. 이 분석법으로 유전형 분석이 가능한 유전자는 CYP2C19, CYP3A4, CYP3A5, CYP4F2, MDR1 등이 있다.
 |
 |
| < PCR > |
< PCR-RFLP 결과> |
7. Chip 분석 기술
Microarray 분석방법은 DNA, 단백질, 항체 또는 세포 등의 생체물질들을 유리, 실리콘, 고분자 등의 고체기질 위에 고밀도로 집적화한 생체정보 감지소자 극미량의 시료를 초고속으로 분석하는데 매우 적절한 기술로 유전자 발현양상, 유전자 결함, 단백질 분포 등의 생물학적 정보를 얻거나 생화학적 동정 및 반응 속도 또는 정보처리 속도를 높이는 방법이다.
유전형 분석에 사용되는 방법은 합성된 single strand oligonucleotied를 화학적인 알데하이드기나 아민기가 코팅되어 있는 유리슬라이드 위에 고정화 시켜 DNA Chip을 준비하고, 조직이나 세포에서 추출, 증폭된 유전물질에 형광물질 Cy3(532nm)나 Cy5(635nm)를 붙여 준비된 DNA Chip과 hybridization 반응을 통해 특정위치의 oligonucleotied와 결합하게 하여 Microarray Scanner로 광학적인 신호를 측정하여 SNP(Sing Nucleotide Polymorphism)을 분석한다. Microarray 분석방법은 한 샘플에서 많은 종류의 변이를 분석할 때 유용한 방법으로 각각의 변이를 따로 확인하는 것에 비해 비용을 절감할 수 있다.
 |
 |
 |
| < Chip 장비 > |
< Chip 장비 > |
< Microarray 결과 > |
|

